RANSACとは
RANSACとは、RANdom SAmple Consensusの略で、ロバストなモデル推定のアルゴリズムである。
(ロバスト推定とは与えられた観測値に外れ値が含まれているとして、その影響を抑えることを目的とした推定)
処理の手順は、
- ①データからランダムに数点を抽出する
- ②誤差がしきい値以下であればそのモデルを正しいモデル候補に追加する
- ③抽出したデータを元に最小二乗法などで仮のモデルを推定し、そのモデルと全体の誤差を求める
- ④仮のモデルからの距離がしきい値以下のデータ点をインライア(inlier: 外れ値でないデータ)とする
- ⑤インライアの数がしきい値以上、または既定の回数に達するまで上記の手順を繰り返す
下にOpen3DのRANSACアルゴリズム抜粋している
registration_ransac_based_on_feature_matching関数 o3d.pipelines.registration.registration_ransac_based_on_feature_matchingは、Open3Dライブラリの一部で、RANSAC(RANdom SAmple Consensus)アルゴリズムを使用して特徴マッチングに基づく3Dレジストレーション(つまり、異なる視点からの3Dスキャンを一緒に整列させるプロセス)を行います。特徴マッチングに基づくRANSACアルゴリズムを使用して、2つの点群間の最適なリジッド変換(回転と並進)を見つけます。この関数の出力は、変換行列とフィットネススコアであり、フィットネススコアがある閾値を超えるまでRANSACアルゴリズムが繰り返されます。
個々の引数は、
pcd1_d,pcd2_d: これらは比較するダウンサンプリングされた2つの点群データを表します。pcd1_f,pcd2_f: これらは上記の点群データに対応する特徴量を表します。mutual_filter: これは相互フィルタリングを有効にするかどうかを指定します。Trueに設定すると、相互に最も近い対応関係のみが保持されます。max_correspondence_distance: これは対応点間の最大距離を指定します。この値より大きい距離の対応関係は無視されます。estimation_method: これは変換行列の推定方法を指定します。ここではest_ptp(point-to-point)が使用されています。ransac_n: これはRANSACアルゴリズムで一度にサンプリングする点の数を指定します。checkers: これは終了条件をチェックするための関数のリストを指定します。criteria: これはRANSACの終了条件を指定します。
#Multi-times RANSAC Matching
iternum = 0
fitness1 = 0
thresold = 0.6 #fitness
while fitness1 < thresold:
result1 = o3d.pipelines.registration.registration_ransac_based_on_feature_matching(
pcd1_d, pcd2_d,
pcd1_f, pcd2_f,
mutual_filter= True,
max_correspondence_distance= size * 2, #2
estimation_method= est_ptp,
ransac_n = 4,
checkers = checker,
criteria = criteria
)
fitness1 = result1.fitness
#print("result1", result1)
print("fitness", fitness1, "iternum", iternum)
iternum += 1
#if iternum > 5:
#break
ICPとは
ICPとは、Iterative Closest Point(ICP)は、3D点群の注目する点に最も近い点を求めるアルゴリズムです。このアルゴリズムは、2つの点群の間で最適な位置関係を見つけるために使用されます。
処理の手順は、
- ①初期位置として、両方の点群を重心に配置します。
- ②参照点群から最も近い点を探すことにより、注目する点群を更新します。
- ③参照点群と注目する点群の間で対応する点を定義します。
- ④対応する点を使用して、注目する点群を回転、平行移動することで最適な位置を見つけます。
- ⑤更新された位置に基づいて、操作を繰り返します。
- ⑥ある停止条件(例えば変化量が一定以下になったとき)が満たされるまで繰り返します。
registration_icp関数o3d.pipelines.registration.registration_icpは、Open3Dライブラリの一部で、ICP(Iterative Closest Point)アルゴリズムを使用して3Dレジストレーション(つまり、異なる視点からの3Dスキャンを一緒に整列させるプロセス)を行います。ICPアルゴリズムを使用して、2つの点群間の最適なリジッド変換(回転と並進)を見つけます。この変換は、一方の点群を他方に整列させるために使用されます。
個々の引数は、
pcd1,pcd2: これらは比較する2つの点群データを表します。size: これは対応点間の最大距離を指定します。この値より大きい距離の対応関係は無視されます。result1.transformation: これは初期変換行列を指定します。この変換は、ICPアルゴリズムが開始する前にpcd1に適用されます。この変換行列は通常、前のレジストレーション手順(この場合はRANSAC)から得られます。est_ptpln: これは変換行列の推定方法を指定します。ここではest_ptpln(point-to-plane)が使用されています。
#Modifying slightly by ICP
result2 = o3d.pipelines.registration.registration_icp(pcd1, pcd2, size, result1.transformation, est_ptpln)RANSACとICPの使い分け
RANSAC(RANdom SAmple Consensus)とICP(Iterative Closest Point)は、どちらも3Dレジストレーションのためのアルゴリズムですが、それぞれ異なる状況と目的に適しています。
RANSACは、大量のデータの中からモデルを推定するためのロバストな方法です。このアルゴリズムは、データの一部が大きなノイズや外れ値を含んでいても、正確なモデルを推定することができます。しかし、RANSACはランダムなサンプリングに依存しているため、結果の再現性が保証されません。また、最適な結果を得るためには、適切なパラメータ設定が必要です。
一方、ICPは、2つの点群を最適に一致させるための反復的な方法です。ICPは、初期の対応関係が既にある程度正確であると仮定しています。そのため、ICPは微調整やリファインメントによく使用されます。しかし、ICPは局所的な最適解に陥りやすく、初期対応が大きくずれていると、全体的な最適解を見つけることが難しくなります。
したがって、RANSACとICPのどちらが「性能が良い」かは、具体的な状況と目的によります。一般的には、初期対応が不確かな場合や大きなノイズが存在する場合はRANSACを、精密な一致が必要な場合や初期対応が既にある程度正確な場合はICPを使用すると良いでしょう。また、両者を組み合わせて使用することも一般的です。例えば、RANSACを使用して大まかな対応関係を見つけ、その結果を初期値としてICPを使用して精密な一致を行う、といった方法があります。今回はそういった意味で、RANSACを用いてからICPを用いている。
実行結果

入力画像である位置姿勢が異なる4つのラビットのモデルである。

モデルが初期値としてある程度同じ位置姿勢の場合はよくフィッティング出来ているという結果が得られた。一方で、一つのモデルだけ位置と姿勢が他とだいぶ異なるモデルを追加してみよう。

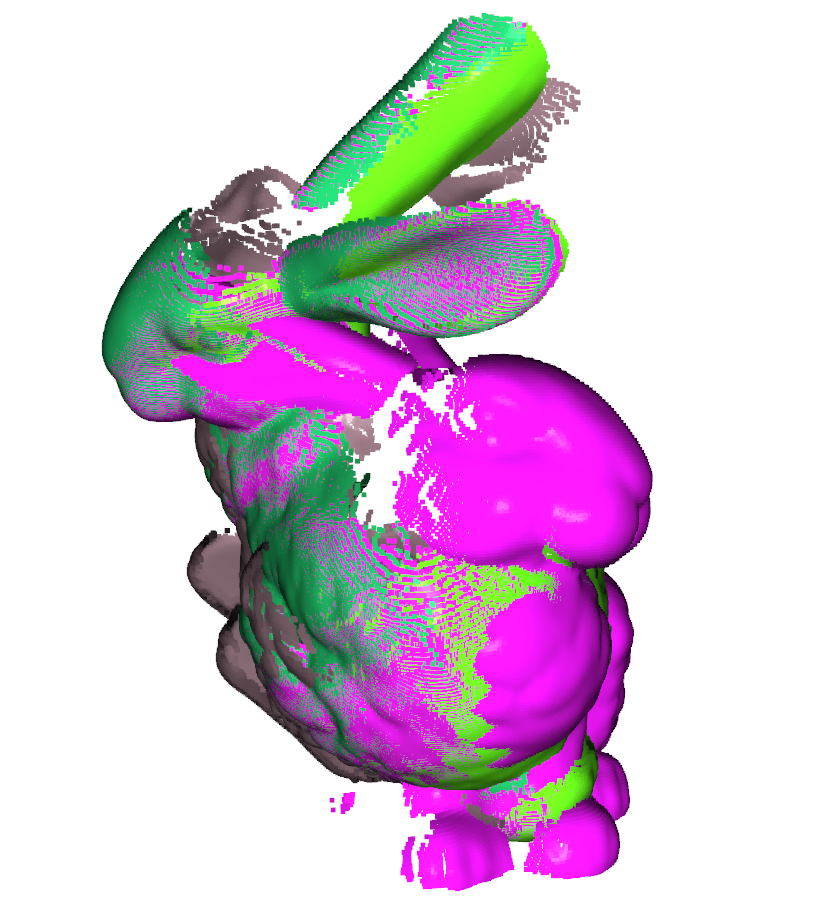
結果は1つのモデルだけ異なる姿勢を向いている。現時点ではこの問題を解決するところまで作りこんでいないが今後の課題としておこう。
ソースコード
import sys
sys.path.append("../..") # Open3D/build/lib/ へのパス
import numpy as np
import open3d as o3d
def register(pcd1, pcd2, size):
# Position alignment between two point clouds
kdt_n = o3d.geometry.KDTreeSearchParamHybrid(radius=size, max_nn=50)#50
kdt_f = o3d.geometry.KDTreeSearchParamHybrid(radius=size *10, max_nn=50)#50
# Downsampling
pcd1_d = o3d.geometry.PointCloud.voxel_down_sample(pcd1, size)
pcd2_d = o3d.geometry.PointCloud.voxel_down_sample(pcd2, size)
o3d.geometry.PointCloud.estimate_normals(pcd1_d, kdt_n)
o3d.geometry.PointCloud.estimate_normals(pcd2_d, kdt_n)
#o3d.visualization.draw_geometries([pcd1_d], "down sampling")
#Feature amount cal
pcd1_f = o3d.pipelines.registration.compute_fpfh_feature(pcd1_d, kdt_f)
pcd2_f = o3d.pipelines.registration.compute_fpfh_feature(pcd2_d, kdt_f)
#Preparation
checker = [o3d.pipelines.registration.CorrespondenceCheckerBasedOnEdgeLength(0.9),
o3d.pipelines.registration.CorrespondenceCheckerBasedOnDistance(size * 0.2)]
est_ptp = o3d.pipelines.registration.TransformationEstimationPointToPoint()
est_ptpln = o3d.pipelines.registration.TransformationEstimationPointToPlane()
criteria = o3d.pipelines.registration.RANSACConvergenceCriteria(max_iteration=5000000, confidence=0.99) #max_validation=500
#Multi-times RANSAC Matching
iternum = 0
fitness1 = 0
thresold = 0.6 #fitness
while fitness1 < thresold:
result1 = o3d.pipelines.registration.registration_ransac_based_on_feature_matching(
pcd1_d, pcd2_d,
pcd1_f, pcd2_f,
mutual_filter= True,
max_correspondence_distance= size * 2, #2
estimation_method= est_ptp,
ransac_n = 4,
checkers = checker,
criteria = criteria
)
fitness1 = result1.fitness
#print("result1", result1)
print("fitness", fitness1, "iternum", iternum)
iternum += 1
#if iternum > 5:
#break
#Modifying slightly by ICP
result2 = o3d.pipelines.registration.registration_icp(pcd1, pcd2, size, result1.transformation, est_ptpln)
return result2.transformation
def merge(pcds):
#Multi point clouds to single point clouds
all_pcds = []
for pcd in pcds:
all_pcds.append(np.asarray(pcd.points))
merge_pcd = o3d.geometry.PointCloud()
merge_pcd.points = o3d.utility.Vector3dVector(np.vstack(all_pcds))
return merge_pcd
#in-place coloring and adding normal
def add_color_normal(pcd):
pcd.paint_uniform_color(np.random.rand(3))
size = np.abs((pcd.get_max_bound() - pcd.get_min_bound())).max() / 30
kdt_n = o3d.geometry.KDTreeSearchParamHybrid(radius=size, max_nn=50)
#o3d.geometry.PointCloud.estimate_normals((pcd, kdt_n))
o3d.geometry.PointCloud.estimate_normals(pcd, kdt_n)
def load_pcds(pcd_files):
pcds = []
for f in pcd_files:
pcd = o3d.io.read_point_cloud(f)
#o3d.visualization.draw_geometries([pcd])
add_color_normal(pcd)
pcds.append(pcd)
return pcds
def align_pcds(pcds, size):
pose_graph = o3d.pipelines.registration.PoseGraph()
accum_pose = np.identity(4)
pose_graph.nodes.append(o3d.pipelines.registration.PoseGraphNode(accum_pose))
n_pcds = len(pcds)
for source_id in range(n_pcds):
print("source_id", source_id)
for target_id in range(source_id + 1, n_pcds):
print("target_id", target_id)
source = pcds[source_id]
target = pcds[target_id]
trans = register(source, target, size)
GTG_mat = o3d.pipelines.registration.get_information_matrix_from_point_clouds(
source, target, size, trans
)
if target_id == source_id + 1:
accum_pose = trans @ accum_pose
pose_graph.nodes.append(o3d.pipelines.registration.PoseGraphNode(np.linalg.inv(accum_pose)))
#print("source_id, target_id, trans, GTG_mat",source_id, target_id, trans, GTG_mat)
pose_graph.edges.append(o3d.pipelines.registration.PoseGraphEdge(
source_id, target_id, trans, GTG_mat, uncertain = True
))
solver = o3d.pipelines.registration.GlobalOptimizationLevenbergMarquardt()
criteria = o3d.pipelines.registration.GlobalOptimizationConvergenceCriteria()
option = o3d.pipelines.registration.GlobalOptimizationOption(
max_correspondence_distance = size/10,
edge_prune_threshold = size/10,
reference_node = 0
)
o3d.pipelines.registration.global_optimization(
pose_graph, method=solver, criteria=criteria, option=option)
for pcd_id in range(n_pcds):
trans = pose_graph.nodes[pcd_id].pose
pcds[pcd_id].transform(trans)
return pcds
pcds = load_pcds([r"C:\Users\chin.ply",
r"C:\Users\bun000.ply",
r"C:\Users\bun045.ply",
r"C:\Users\bun315.ply"
])
o3d.visualization.draw_geometries(pcds, "input pcds")
size = np.abs((pcds[0].get_max_bound() - pcds[0].get_min_bound())).max() / 30
pcd_aligned = align_pcds(pcds, size)
o3d.visualization.draw_geometries(pcd_aligned, "aligned")
pcd_merge = merge(pcd_aligned)
add_color_normal(pcd_merge)
o3d.visualization.draw_geometries([pcd_merge], "merged")
コメント